
引言#
最近要用nnUNet来处理医学图像,准确来说叫图像分割。简单来说就是训练一个模型,使之给定他一个图片,他能够将图片的血管分割出来。此次使用的工具是nnUNet ↗,nnUNet是一个自适应的深度学习框架,专为医学图像分割任务设计。他的最大的特点就是自动化,他自动训练训练集,自动预训练,自动评判。简而言之,他就是一个功能多样且强大,封装好的医学图像分割工具。
所以本次博客主要记录一下如何使用nnUNet进行训练。
配置#
- mac m1 pro 16 + 512
配置环境#
一开始我们要clone nnUNet的仓库到我们的本地,当然也可以不用,要是没有需求修改他们的模型就不用。 但这里我clone了
git clone https://github.com/MIC-DKFZ/nnUNet.git
cd nnUNet然后建议用conda或者其他的来管理python包这里要安装的还挺多的
pip install -e .是用于在当前目录下以“可编辑模式”(editable mode)安装 Python 包的命令。具体来说,这个命令会查找当前目录中的 setup.py(或者 pyproject.toml),并将包安装到 Python 环境中,但不是把文件复制过去,而是建立一个指向源代码的链接。这意味着当你修改源码时,无需重新安装,修改会立即生效,非常适合开发调试阶段。
数据处理#
我一开始拿到的数据集其实并不是按照nnUNet标准要求的数据格式来存放的。以下是我一开始数据集的结构图。我一开始有两个数据集,每个数据集的格式都是如此
/path/to/your_dataset_1/
|——crop
|——crop_test
|——crop_train
| |——0
| |——image.png
| |——label.png
| |——mask.png
| |——1
| …
|——original - original:存放原始图片,用于以防万一后续检查
- crop:切割后的图片,就是将原始图片进行裁切,放大我们想要关注的区域
- crop_train:他和crop_test平分crop中的图片,一半用于训练,一半用来训练完之后的检验。然后这个数据有0,1,2, …个文件夹,里面存的image.png, label.png, mask.png
- image.png 切割后的图片
- label.png 标签,就是人工标注的分割结果,用于训练和检测
- mask.png 测试的时候防止预测区域外干扰使用的
- crop_test 结构和crop_train一样
但可惜的是,nnUNet支持的格式并不是这种, 数据集必须位于 nnUNet_raw 文件夹中(你可以在安装 nnU-Net 时定义该文件夹,或在每次运行 nnU-Net 命令时导出/设置该路径)。每个分割数据集都作为一个独立的“Dataset”存储,并关联一个数据集 ID(一个三位整数)和一个你自定义的数据集名称。例如,Dataset005_Prostate 的名称为 “Prostate”,其数据集 ID 为 5。数据集在 nnUNet_raw 文件夹中的组织结构如下:
nnUNet_raw/
├── Dataset001_BrainTumour
├── Dataset002_Heart
├── Dataset003_Liver
├── Dataset004_Hippocampus
├── Dataset005_Prostate
├── ...在每个数据集文件夹内,期望的结构如下:
Dataset001_BrainTumour/
├── dataset.json
├── imagesTr
├── imagesTs # 可选
└── labelsTr- imagesTr:存放训练样本的图像。nnU-Net 会基于这些数据进行管道配置、交叉验证训练、后处理以及寻找最佳集成策略。
- imagesTs(可选):存放测试样本的图像。nnU-Net 不会使用它们,仅作为一个便于存储的目录,这是 MSD 文件夹结构的遗留部分。
- labelsTr:存放训练样本的真实分割图。
- dataset.json:包含数据集的元数据信息。
上述方案将产生如下文件夹结构。以下以 MSD 的第一个数据集 BrainTumour 为例,该数据集有四个输入通道:FLAIR(0000)、T1w(0001)、T1gd(0002)和 T2w(0003)。注意,imagesTs 文件夹为可选。
nnUNet_raw/Dataset001_BrainTumour/
├── dataset.json
├── imagesTr
│ ├── BRATS_001_0000.nii.gz
│ ├── BRATS_001_0001.nii.gz
│ ├── BRATS_001_0002.nii.gz
│ ├── BRATS_001_0003.nii.gz
│ ├── BRATS_002_0000.nii.gz
│ ├── BRATS_002_0001.nii.gz
│ ├── BRATS_002_0002.nii.gz
│ ├── BRATS_002_0003.nii.gz
│ ├── ...
├── imagesTs
│ ├── BRATS_485_0000.nii.gz
│ ├── BRATS_485_0001.nii.gz
│ ├── BRATS_485_0002.nii.gz
│ ├── BRATS_485_0003.nii.gz
│ ├── BRATS_486_0000.nii.gz
│ ├── BRATS_486_0001.nii.gz
│ ├── BRATS_486_0002.nii.gz
│ ├── BRATS_486_0003.nii.gz
│ ├── ...
└── labelsTr
├── BRATS_001.nii.gz
├── BRATS_002.nii.gz
├── ...下面是 MSD 第二个数据集的另一个示例,该数据集只有一个输入通道:
nnUNet_raw/Dataset002_Heart/
├── dataset.json
├── imagesTr
│ ├── la_003_0000.nii.gz
│ ├── la_004_0000.nii.gz
│ ├── ...
├── imagesTs
│ ├── la_001_0000.nii.gz
│ ├── la_002_0000.nii.gz
│ ├── ...
└── labelsTr
├── la_003.nii.gz
├── la_004.nii.gz
├── ...注意:nnUNet version 2 已经不止支持nii.gz格式,包括png等的很多格式都已经支持,具体可以看他们的官方文档 ↗
所以我们要做的就是将数据集中的crop_train中的训练图片和label分出来,存到对应的文件夹中。此外我们还要根据我们的数据写dataset.json文件。
脚本如下:
import os
import shutil
from batchgenerators.utilities.file_and_folder_operations import maybe_mkdir_p, save_json
from PIL import Image
import numpy as np
def organize_dataset(input_dir, output_dir, is_label=False):
for case in os.listdir(input_dir):
case_dir = os.path.join(input_dir, case)
if os.path.isdir(case_dir):
image_file = os.path.join(case_dir, 'image.png')
label_file = os.path.join(case_dir, 'label.png') if is_label else None
# Copy image to output directory
if label_file is None:
img = Image.open(image_file).convert("L")
img.save(os.path.join(output_dir, f'{case}_0000.png'))
if is_label:
label_image = Image.open(label_file)
label_array = np.array(label_image)
label_array[label_array == 255] = 1
converted_label_image = Image.fromarray(label_array)
converted_label_image.save(os.path.join(output_dir, f'{case}.png'))
def main():
# base_dir = '/Path/to/your/dataset/crop_train'
base_dir = '/Path/to/your/dataset/crop_train'
# test_dir = '/Path/to/your/dataset/crop_test'
test_dir = '/Path/to/your/dataset/crop_test'
# nnunet_raw_dir = '/Path/to/nnUNet_raw/Dataset001_****'
nnunet_raw_dir = '/Path/to/nnUNet_raw/Dataset001_****'
imagesTr_dir = os.path.join(nnunet_raw_dir, 'imagesTr')
labelsTr_dir = os.path.join(nnunet_raw_dir, 'labelsTr')
imagesTs_dir = os.path.join(nnunet_raw_dir, 'imagesTs')
maybe_mkdir_p(imagesTr_dir)
maybe_mkdir_p(labelsTr_dir)
maybe_mkdir_p(imagesTs_dir)
organize_dataset(base_dir, imagesTr_dir)
organize_dataset(base_dir, labelsTr_dir, is_label=True)
organize_dataset(test_dir, imagesTs_dir)
if __name__ == '__main__':
main()值得注意的是,这里面还有一些细节。一开始我们的图片都是有色的,意味着我们的图片每个像素都有三个参数RGB,在nnUNet中就表示多通道,但是我训练的时候是按单通道来训练,所以我就将这个图片转化为黑白的。
| 原图 | 处理后 |
|---|---|
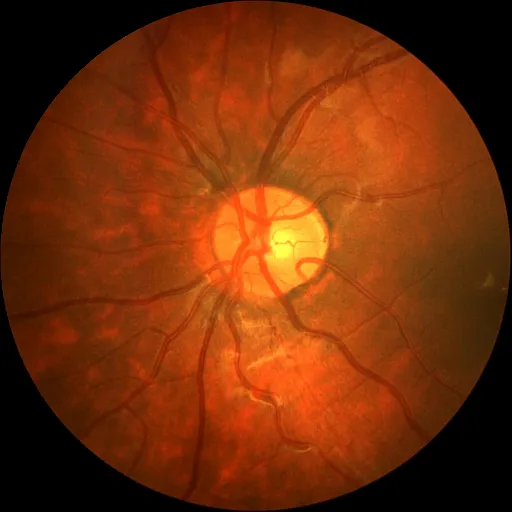 | 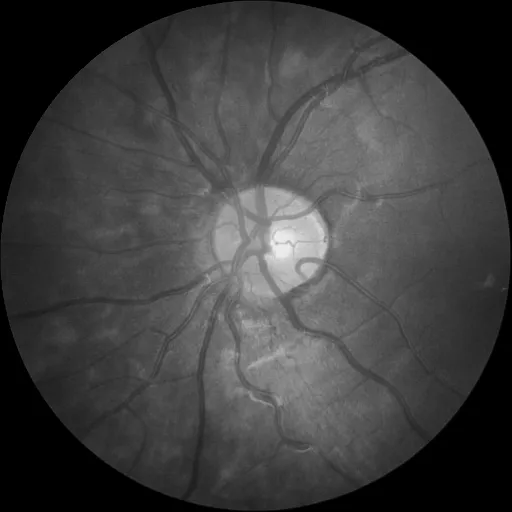 |
其次label的图像也要处理,我的数据集中,label是黑白的,意味着图片的数组就是[0,255],0表示黑色,255表示白色。在nnUNet中,0被强制表示为背景,也就是背景必须为黑,然后其他的参数必须要连续,所以我的图片的数组就必须是[0,1],所以这个脚本将图片中的255全都改成0。反映出来的结构就是,每一个处理后的图片都几乎是全黑的,因为0,1这两个灰度很接近,所以整张图几乎就是全黑的。但是没有关系,我们人眼分辨出来计算机可以,这满足了nnUNet的格式。到最后我们只用再把1改成255就可以分辨了。
| 原图 | 处理后 |
|---|---|
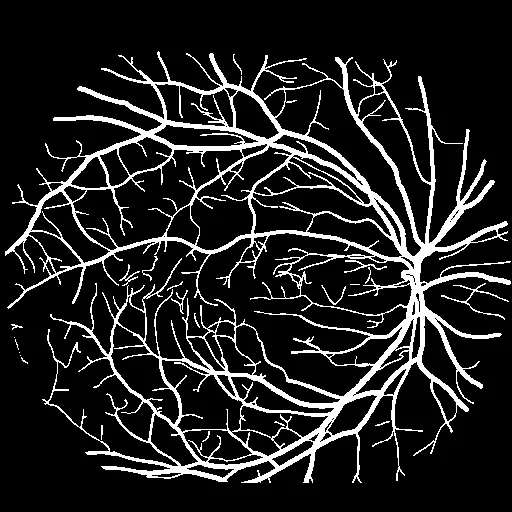 |  |
分类完这些图片之后,就是data.json文件,这个文件的作用就是告诉模型一些元数据。
元数据(Metadata)就是描述数据的一组信息,它提供了关于数据本身的背景、结构、属性等为模型训练提供了“关于数据的指南”,使得整个训练流程——从预处理、数据增强、网络架构设计到最终的推理和后处理——都能基于具体数据集的特点自动调整,从而提高模型的适用性和性能。
以下是我的json文件
{
"channel_names": {
"0": "CT"
},
"labels": {
"background": 0,
"lesion": 1
},
"numTraining": 14,
"file_ending": ".png"
}- channel:就代表输入有几个通道,我只有一个就只有0
- labels:就代表label的结构,0表示背景,1表示白色的部分
- numTraining:表示训练图像有多少个
- file_ending:表示图片的格式
将json文件放到指定位置之后数据处理部分就完成
预训练#
预训练通过在通用数据上建立良好的基础,使得在面对具体任务时,模型能够更快、更高效地学习并达到较好的表现。
运行预处理的最简单方式为:
nnUNetv2_plan_and_preprocess -d DATASET_ID --verify_dataset_integrity其中 DATASET_ID 即数据集的编号。我们建议首次运行时总是加上 --verify_dataset_integrity 选项,以检查一些最常见的错误来源!
- DATASET_ID:就是一开数据命名的,要是Dataset001,就填1
这一个过程很快,结束后就再 nnUNet_preprocessed中看到结果
训练#
这一步开始我们就正式训练,
nnUNetv2_train DATASET_NAME_OR_ID UNET_CONFIGURATION FOLD [其他选项, 详见 -h]- DATASET_NAME_OR_ID:就是数据集编号,1,2,3这些
- UNET_CONFIGURATION :是标识所请求 U-Net 配置的字符串(默认包括 2d、3d_fullres、3d_lowres、3d_cascade_lowres)我训练的2d的我就选2d
- FOLD:选第几个fold当作训练时的验证集,默认他总共有5折
- -device (cpu,gpu,mps):这里就是训练的设备,mac m1,2可以用mps
- -tr nUNetTrainer_XXepochs:这里就是总共训练几轮,XX是轮数,默认是1000,他可以有1,10,20,50,这些参数,可以去/nnUNet/nnunetv2/training/nnUNetTrainer/variants/training_length/nnUNetTrainer_Xepochs.py里面看有什么选项
这些还挺重要的,别像我一开始mac用cpu加速,然后epochs默认1000次,算了一下大概要3个月,才能训完一个只有50张图片的模型,然后我的电脑还嘎嘎烫。后来我用了mps以及50 epochs,大概要5个小时。
训练过程中他会有一些反馈,告诉你当前是第几轮了。
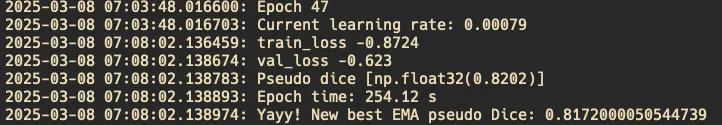
比如图中
- Epoch:就是第几轮的意思,我这是47轮
- current learning rate:就是当前的学习率,一般来说刚开始训练的时候,这个会比较大,简单来说就是一开始像梯度下降的地方快速前进,随着训练到后面,你越接近极小值,你的步伐应该越来越小,否则会出现接近极小值的那部分仍然有很多的抖动。
- train_loss:这是 训练集上的损失(loss),表示模型在训练数据上的误差大小。
- val_loss:这是 验证集上的损失,衡量模型在未见过的数据上的表现。
- Pseudo Dice:是 nnU-Net 计算的 Dice 分数,这具体含义我还不了解
- EMA: 指数滑动平均(EMA, Exponential Moving Average) Dice 分数的历史最佳值。
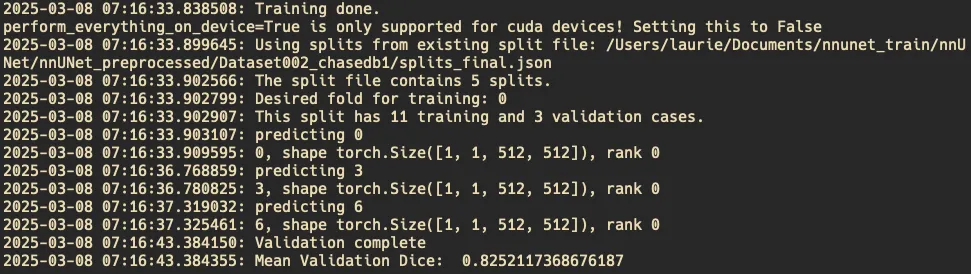
当你训练完成之后就会是这样,这里我用fold 0 当作验证集,意思就是我50个数据被分为5 个fold
用其中的1个当作验证集,其他4个当作训练集。正常来说,我们应该要循环训练所有 fold(通常 5 折交叉验证)意思就是让每一个fold当作一次验证集,最后平均这些结果。但是我的电脑训练一次都已经费劲了,所以我这里只选择了一个fold。后面的数据就是用验证集检验的结果,最后就是validation complets,以及Mean Validation Dice(平均 Dice 系数)= 0.8252,另一个数据集是0.7089
预测#
训练完成之后就让模型处理一些图片
nnUNetv2_predict -i INPUT_FOLDER -o OUTPUT_FOLDER -d DATASET_NAME_OR_ID -c CONFIGURATION --save_probabilities- INPUT_FOLDER: 输入文件路径
- OUTPUT_FOLDER:输出文件路径
- DATASET_NAME_OR_ID:跟之前一样,数据集名字
- CONFIGURATION:2d,3d还是别的
注意:我只用了fold 0作为数据集,但nnUNet的预测会想要用5 fold的数据来预测,所以-f 0 就是让他使用fold 0 来预测 -device mps 就是用mac的架构来预测,默认是cuda
nnUNetv2_predict -i ./nnUNet_raw/Dataset002_chasedb1/imagesTs -o ./nnUNet_raw/Dataset002_chasedb1/output -d 2 -c 2d --save_probabilities -f 0 -device mps运行完之后就可以在对应文件夹中看到分割出来的图片。但注意,输出的图像还是[0,1]的,输出的就是全黑的图片,后面再用脚本将它转化回来。
import os
import numpy as np
from PIL import Image
# 设置你的文件夹路径
folder = "Path/to/your/result"
for filename in os.listdir(folder):
if filename.lower().endswith('.png'):
filepath = os.path.join(folder, filename)
img = Image.open(filepath)
# 如果图片不是RGB模式,可先转换:img = img.convert("RGB")
img_array = np.array(img)
# 将所有像素值为1的元素替换为255
img_array[img_array == 1] = 255
# 转换回图片对象并保存覆盖原文件
new_img = Image.fromarray(img_array)
new_img.save(filepath)这样就可以看到结果
| 预测图像 | 分割图像 |
|---|---|
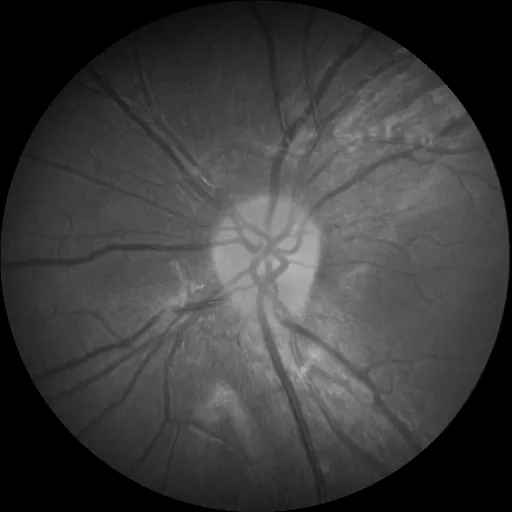 | 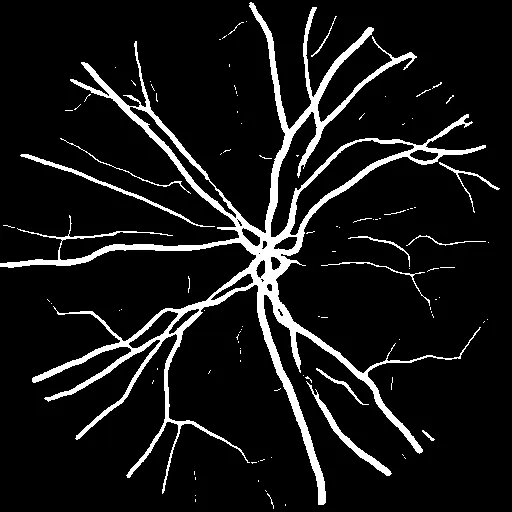 |
小结#

感觉做这个也不容易,我的所有核都跑满了,训练结果不能说很好吧,但是受限于设备这些,我也不可能一直用我的电脑训练,想要更好的结果那就epochs再多点,5个fold全都跑完,在cuda上跑,应该会有不错的效果吧。