
像素级自监督 SAM IEEE Trans 2023 论文笔记
SAM - Self-supervised Learning of Pixel-wise Anatomical Embeddings in Radiological Images
前言#

这篇文章的主要贡献就是在医学图像领域提出一个像素级的自监督解剖嵌入学习框架,主要采用coarse-to-fine结构以及文章中提出的负样本采样策略。
然后在多个任务表现良好,比如lesion matching(病灶匹配),image Registration(图片配准)。病灶配准就是一开始拍摄的图片中可能有个病变,然后一段时间之后再拍一张照片,就能定位原来的那个病变在哪里。image Registration就是把两幅或多幅不同来源、不同时间、不同角度的图像空间上对齐,使它们的同一结构或内容能够准确重合。
Coarse-to-Fine Network Architecture#
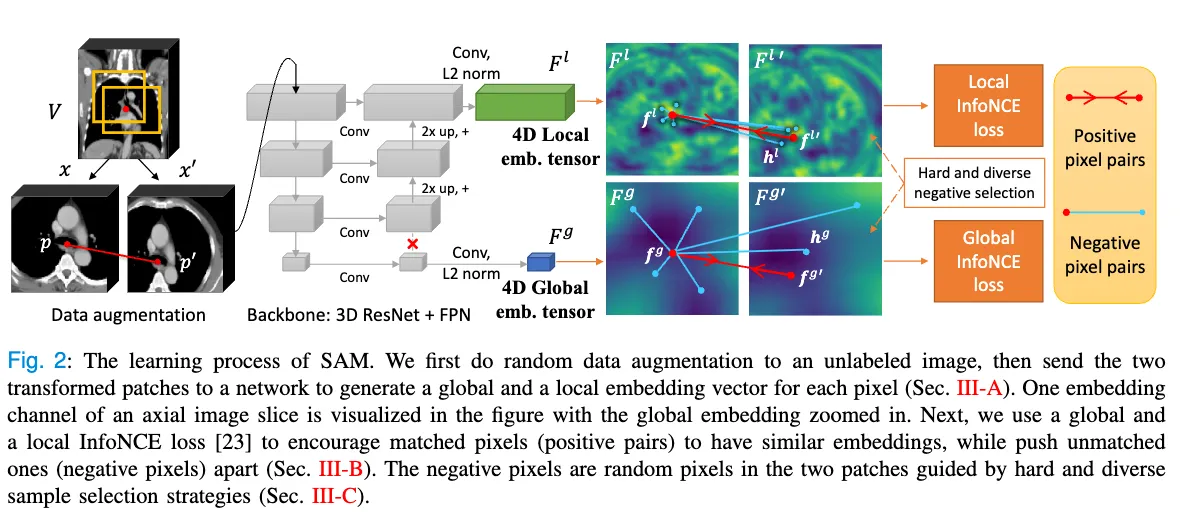
这是文章中提出来的框架,主要用的是ResNet结合魔改过的FPN。补充一下FPN的背景
FPN#
可以看这里 ↗
SAM#
这个架构的主要流程就是对于一张3D的CT图,我从中随机取两个随机大小的patch,然后将这两个patch放大到同一尺寸,分别为 。然后这两个patch就输入进3D的ResNet中,然后经过FPN得到最上层的特征图,以及最下层的特征图经过3*3的卷积和L2 正则化之后的到Global emb tensor 和Local emb tensor 。他们是128通道,也就是每一个像素对应一个128维大小的特征向量。然后就可以得到两个patch分别的local 和 global emb tensor。
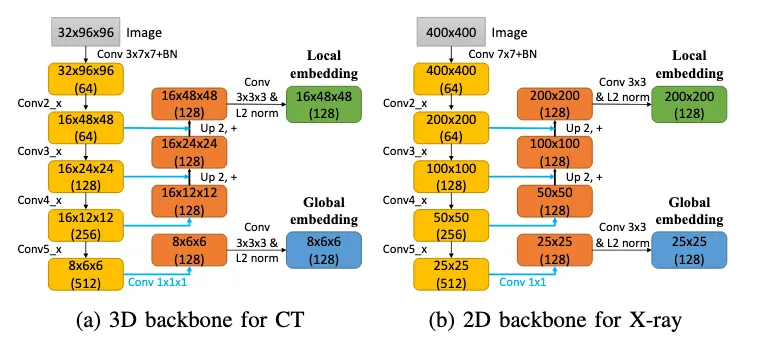 与一开始的FPN不同,他的右边不是从最上面的特征往下的,他切断了顶层,从倒数第二层开始往下。还有他没有用到1*1的卷积核,他的通道数都是设计好的,不需要1*1的卷积核来改变通道数。
与一开始的FPN不同,他的右边不是从最上面的特征往下的,他切断了顶层,从倒数第二层开始往下。还有他没有用到1*1的卷积核,他的通道数都是设计好的,不需要1*1的卷积核来改变通道数。
Pixel Pairs and Loss Function#
然后我们随机取两个patch中重叠部分的同一个像素作为正样本对,分别为 他们对应的特征向量为。然后选 个作为负样本对,用InfoNCE来作为损失函数。
还有值得注意的是,Local 和 Global 分别计算损失函数,然后分别以和 作为锚点(anchor)再计算损失函数(上式中分母那里用的是为锚点,把他改成就是为锚点),因为对称。所以最终的损失函数应该有4部分。
Hard and Diverse Negative Sampling#
这部分就是怎么选他的负样本对,为了模型能学到更多的东西,文章提出他的策略。
对于Global部分,我们用 和, 做卷积得到余弦相似度,得到的similarity map为 和 然后我们选个排除 , 最相似的像素作为hard negatives 。然后为了多样化,还会从同一个training batch中的不同path的随机个像素添加进负样本对中。
对于Local部分,他同样计算从,中计算 similarity map为 和 ,然后他将global 的similarity map 和 上采样到 和 大小,然后直接相加得到combined similarity map 和 ,然后先从中选 个分数最高的,然后才从中挑个作为最终负样本对。这个随机过程,是为了避免所有hard negative都扎堆在某个局部——即使相似,也能让每批hard negative点多样化,避免模型过拟合局部微差。
Application: Anatomical Point Matching#
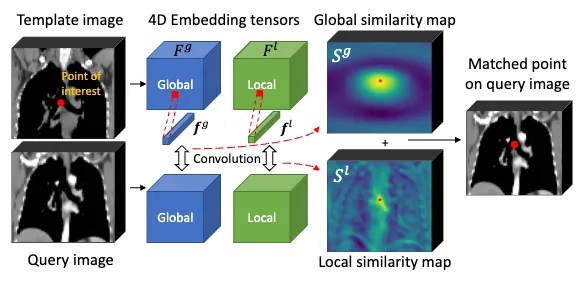 这部分就是讲他怎么用在lesion matching(病灶匹配),一开始给个样本图,然后计算出一开始病灶像素的Local 和 Global的向量,然后同时和后面的图片的tensor做卷积得到相似图,然后结合Local和Gobal的相似图找到后来的病灶位置
这部分就是讲他怎么用在lesion matching(病灶匹配),一开始给个样本图,然后计算出一开始病灶像素的Local 和 Global的向量,然后同时和后面的图片的tensor做卷积得到相似图,然后结合Local和Gobal的相似图找到后来的病灶位置
Application: Enhancing Image Registration#
这部分讲的是怎么用于image Registration(图片配准)。
1. SAM-affine:稀疏点仿射配准#
流程
- 假设有两幅CT:A(固定影像)、B(要对齐的移动影像)。
- 你在A上均匀采样一些网格点(稀疏,比如只采几百/几千个点,而不是全部体素),这些点覆盖A上的各个解剖部位。
- 把网格点外(身体外)的点丢弃。
- 用SAM,把A中每个采样点的embedding,去B中找“特征最相似的点”作为B上的配对。
- 计算这些点对的相似度分数,只有“很可靠的高分”对才留下来。
- 用这些点对,通过最小二乘法直接拟合一个全局仿射变换矩阵(类似“坐标对齐”),实现A、B图的大致空间对正。
2. SAM-coarse:稀疏点粗变形配准#
流程
- 上一步只能做到“整体大致套准”,对器官弯曲、形变等局部结构还无法精准重合。
- 所以,再从配准后的两幅图(A’, B’)中重新采样一批稀疏点(局部更密集一些也可以)。
- 再用SAM嵌入,对齐两图的稀疏点对。
- 根据点对间的空间关系,插值得到一张粗略的变形场(deformation field)(让身体结构能弯曲、拉伸配对)。
3. SAM-VoxelMorph:深度配准精调#
流程
- 经过前面两轮后,A,B已经基本“空间对应”了,但最后精细结构、微小差异依然没法完全对齐。
- 这一步用现有的深度学习配准网络(VoxelMorph)做细调,“吃进去”的输入包含SAM输出的空间相关特征+SAM相似性损失(即每个点对的embedding距离也影响最终效果)。
- SAM相当于做了强力辅助,支持VoxelMorph网络学到更解剖学、判别性更强的变形映射。